Etiologia
O vírus da hepatite B (HBV) é um vírus de ácido desoxirribonucleico (DNA) envelopado, não citopático, hepatotrófico e altamente infeccioso que pertence à família dos hepadnavírus.[22][23] O envelope externo do vírus contém três antígenos de superfície relacionados, sendo o mais abundante deles a proteína S (antígeno de superfície da hepatite B [HBsAg]). O desenvolvimento de imunidade celular e humoral ao HBsAg é uma proteção. Dentro do envelope está o nucleocapsídeo viral, ou core, que contém DNA circular de fita parcialmente dupla (antígeno core da hepatite B [(HBcAg]). Os peptídeos derivados de HBcAg induzem a uma resposta celular imune do hospedeiro crucial contra o HBV. O antígeno E da hepatite B, uma proteína viral da hepatite B, serve de marcador para uma replicação ativa, mas sua função é desconhecida. A proteína X pode ter participação na evolução do carcinoma hepatocelular. A polimerase do DNA tem função de transcriptase reversa na síntese de fitas negativas e positivas do DNA do HBV.[22][23]
Fisiopatologia
O vírus não mata diretamente os hepatócitos.[23] Acredita-se que a resposta imune do hospedeiro aos antígenos virais seja a causa da lesão hepática na infecção por vírus da hepatite B (HBV).[24] A resposta imune celular, e não a resposta imune humoral, parece estar fundamentalmente envolvida na patogênese da doença. Acredita-se que a indução da resposta do linfócito T antígeno específica ocorra quando os linfócitos T do hospedeiro são apresentados aos epítopos virais pelas células apresentadoras de antígenos nos órgãos linfoides. Essas células T específicas para antígenos amadurecem e se expandem e, depois, migram para o fígado. Na infecção aguda por HBV, a maior parte do ácido desoxirribonucleico (DNA) do HBV é depurada dos hepatócitos através de efeitos não citocidas dos subprodutos inflamatórios dos linfócitos T CD8+, estimulados pelos linfócitos T CD4+, principalmente, a gamainterferona e o fator de necrose tumoral alfa. Eles provocam a down-regulation da replicação viral e desencadeiam a lise direta de hepatócitos infectados pelas células T citotóxicas CD8+ específicas para HBV.[25] Em contraste, pessoas com infecção crônica por HBV mostram respostas fracas, infrequentes e restritas às células T específicas para HBV, e a maioria das células mononucleares em fígados de pessoas infectadas cronicamente com HBV não são antígeno inespecíficas.[26]
[Figure caption and citation for the preceding image starts]: Ciclo de vida do vírus da hepatite B (HBV)De Ganem D, Prince AM. Hepatitis B virus infection - natural history and clinical consequences. 2004; 350:1118-1129; usado com permissão [Citation ends].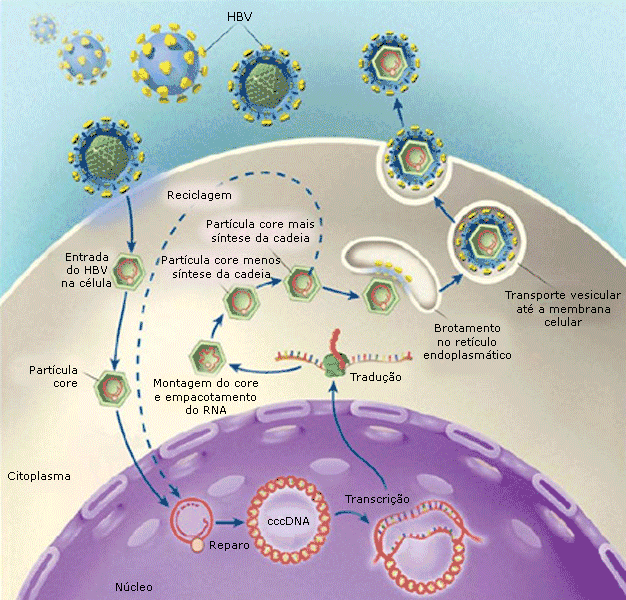
Devido à presença de HBV em locais extra-hepáticos, assim como a presença de DNA circular covalentemente fechado (cccDNA) dentro dos hepatócitos, a erradicação do vírus é uma meta irreal com base nos medicamentos disponíveis atualmente. O DNA circular covalentemente fechado serve de molde para a transcrição do ácido ribonucleico (RNA) mensageiro pré-genômico, uma etapa inicial vital para a replicação do HBV.[27][28][29][30] A presença contínua do cccDNA nos hepatócitos é considerada um marcador da persistência viral. Infelizmente, as terapias atuais não foram efetivas na erradicação do cccDNA e só conseguem reduzir os níveis.[31][32][33][34][35][36][37] Demonstrou-se que a persistência de níveis baixos de cccDNA no núcleo dos hepatócitos está relacionada ao efeito rebote do vírus após a interrupção da terapia. Além disso, a integração do DNA do HBV ao núcleo do hepatócito durante o processo de replicação poderia explicar o aumento do risco de carcinoma hepatocelular. Além disso, a coinfecção com o vírus da hepatite C (HCV) pode aumentar sinergicamente a taxa de fibrose, cirrose e câncer hepatocelular, porque tanto o HBV quanto o HCV podem infectar o mesmo hepatócito independentemente.[38][39][40][41][42]
Classificação
genótipo do HBV
Os genótipos do vírus da hepatite B (HBV) são baseados nos 8% de variação intertípica da sequência completa de nucleotídeos do genoma e são distribuídos geograficamente.[1] Dados mostram que os genótipos do HBV podem ter um papel na progressão da doença hepática relacionada ao HBV e na resposta à terapia com interferona.[2] Estudos demonstraram aumento da soroconversão do anticorpo antiantígeno E do vírus da hepatite B (anti-Hbe) com o genótipo A e o genótipo B, embora um estudo tenha demonstrado que a melhora na soroconversão do anti-Hbe foi limitada apenas ao genótipo A.[3][4][5][6][7] Como melhoras na resposta ao tratamento têm sido observadas no tratamento com interferona e não na terapia com nucleotídeo/nucleosídeo, novas pesquisas seriam úteis antes de recomendar o teste de genótipo na prática clínica e de fazer a correlação com a resposta ao tratamento. Atualmente, a genotipagem não é recomendada para testes de rotina ou acompanhamento de pacientes com infecção crônica por HBV.[2]
A distribuição mundial dos genótipos varia.[8]
O genótipo A é encontrado principalmente nos EUA, no noroeste da Europa e no sudeste da África.
O genótipo A é associado a uma taxa maior de soroconversão de HBeAg com terapia com interferona em comparação com os genótipos B, C e D.
Os genótipos B e C são encontrados principalmente no Sudeste Asiático, China e Japão.
O genótipo B está associado a uma idade menor na soroconversão do HBeAg, à necroinflamação hepática menos ativa, à remissão mais sustentada após a soroconversão do HBeAg, a uma menor taxa de progressão para cirrose e uma menor taxa de carcinoma hepatocelular (CHC), em comparação com o genótipo C.
O genótipo C é o genótipo mais associado à cirrose e ao CHC.
O genótipo D é encontrado principalmente na bacia do Mediterrâneo e em algumas partes da Ásia, mas foi encontrado em todo o mundo.
O genótipo E é encontrado principalmente na África Ocidental e Central.
O genótipo F é encontrado principalmente na América Central e do Sul.
O genótipo G é encontrado principalmente no México.
O genótipo H é encontrado principalmente no México.
Os genótipos I e J também foram identificados.
O uso deste conteúdo está sujeito ao nosso aviso legal